Project Discovery - Niveau supérieur déverrouillé !
Chers capsuliers,
Il est temps de passer au niveau supérieur et de repousser encore plus loin les limites de vos prouesses dans le Project Discovery : les données seront désormais plus complexes, au profit des découvertes de la communauté scientifique ! En qualité de pionniers immortels de New Eden, vous avez d'ores et déjà surpassé les espoirs les plus fous nourris pour ce projet, fondés à l'aune de vos performances exceptionnelles au cours des précédentes mises en œuvre du Project Discovery.
« Nous travaillons depuis 5 ans sur le Project Discovery, et il est impressionnant de constater que même après tout ce temps, l'investissement remarquable de la communauté de joueurs d'EVE dans les efforts de recherche n'a pas faibli. Cette contribution est particulièrement importante et précieuse en ces temps troublés. Merci à tous. » - Attila Szantner, PDG et cofondateur de MMOS.
Lors de la conception et de l'implémentation de cette dernière version du Project Discovery, nous avons été menés à affiner nos connaissances sur la cytométrie en flux et les populations de cellules afin de rendre le contenu attrayant pour les joueurs, tout en fournissant des résultats précis à nos scientifiques. Dans la mesure où 39,6 millions de contributions ont été capitalisées depuis le lancement du projet, nous pouvons dire sans trop nous avancer que les joueurs d'EVE Online ont répondu massivement à l'appel de la lutte contre la COVID-19.
« La participation et la qualité des données recueillies par la communauté d'EVE se sont révélées exceptionnelles et ont dépassé toutes nos attentes. Nous nous réjouissons de pouvoir exploiter cette source unique de savoir pour le bénéfice immédiat de la recherche biomédicale. » - Jérôme Waldispühl, maître de conférences, École d'informatique, Université McGill.
Avec 466 000 échantillons déjà vérifiés et prêts à être utilisés pour la recherche scientifique, nous pouvons affirmer sans risque que ces données, aussi nombreuses que rigoureuses, n'existeraient pas sans les efforts de tous les joueurs d'EVE dévoués à la cause dans le monde entier. Néanmoins, l'heure est venue d'optimiser la finesse des analyses pour faire face à l'afflux ininterrompu d'échantillons.
Lorsque le projet a été introduit, l'objectif principal était de vous apprendre à délimiter des populations uniques sur des graphiques de cytométrie en flux pour transmettre le résultat de vos efforts, les graphiques analysés, aux partenaires de CCP à UNIMORE, MMOS, McGill et à l'Université de la Colombie-Britannique, pour qu'ils puissent exploiter ces données dans l'espoir de mieux comprendre la COVID-19. Des progrès considérables ont été accomplis sur ce front, ce qui est fantastique. Si vous souhaitez en savoir plus sur les avancées réalisées, écoutez ce podcast ou consultez le site web de McGill consacré au projet. Cela dit, nous avions conscience qu'à un moment donné, cette approche devrait être affinée, et le temps est aujourd'hui venu de passer à l'étape supérieure.
« Les joueurs d'EVE Online font un travail exceptionnel. La précision de leurs analyses est très élevée, ce qui démontre l'extraordinaire attention portée sur ce qui n'est pas un simple jeu, mais une lutte réelle contre la COVID. Des joueurs des quatre coins du monde prouvent que la collaboration avec les scientifiques est réellement fructueuse, et qu'ensemble, nous pouvons soulever des montagnes dans un climat plus dramatique et incertain que jamais. » - Dr Andrea Cossarizza, Professeur de Pathologie et d'Immunologie à l'Université de Modène et de Reggio d'Émilie en Italie.
Les participants au Project Discovery ont fourni des données cytométriques inestimables au cours de ces trois derniers mois. Après avoir compilé et analysé vos contributions à l'aune de leur expérience riche de dizaines d'années en laboratoire, nos partenaires scientifiques vous mettent à présent au défi d'établir des graphiques encore plus précis. Cependant, pour mener cette tâche à bien, vous devrez en apprendre davantage sur la cytométrie en flux et les graphiques utilisés, et identifier les points à améliorer.
En ce qui concerne la majorité des graphiques analysés par les capsuliers, les données recueillies relèvent de l'excellence. Tous les échantillons analogues aux graphiques suivants font déjà l'objet d'une étude approfondie. Vous n'avez pas besoin de modifier votre méthode d'analyse pour ces graphiques.
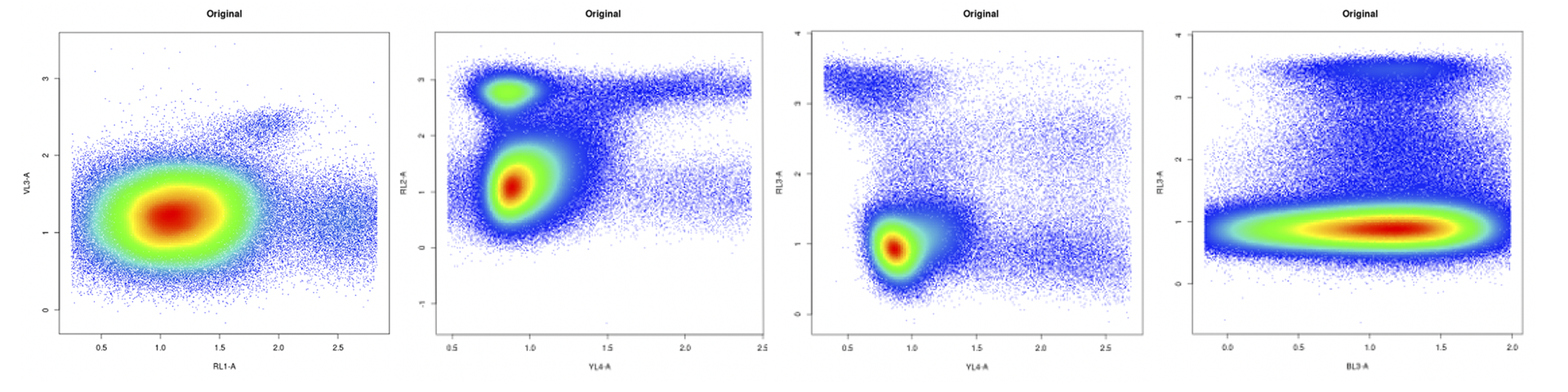
Nous espérons en revanche perfectionner le protocole de délimitation des clusters dans les graphiques ressemblant aux deux modèles suivants, plus complexes à analyser en raison des modalités de conversion des échantillons sanguins et cellulaires réels en graphiques en 2D.
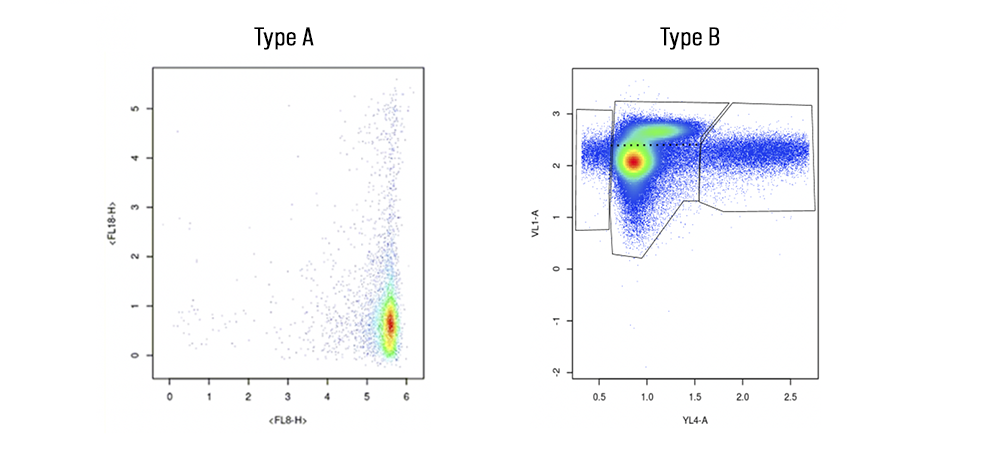
Mieux vaut commencer par distinguer ce qui rend les échantillons de Type A uniques : leurs queues dispersées. Nommée « traces » dans le monde de la cytométrie en flux, la fine bande de cellules qui se dispersent de chaque côté du cluster de haute densité contient de précieuses informations sur la façon dont la COVID-19 affecte les cellules. La présence de traces signifie que les cellules mûrissent lentement et que plusieurs cellules oscillent entre jeunesse et maturité. Pour faire simple, il y a une multiplicité de types de cellules en dépit du nombre restreint de points sur le graphique. La délimitation des populations, c'est-à-dire la localisation des « clusters » de cellules, constitue une ressource inestimable pour comprendre les impacts viraux sur les populations cellulaires. Bien que ces types de graphiques ne semblent pas contenir beaucoup d'informations à propos des cellules, ils regorgent de données essentielles.
Les échantillons de Type A sont également difficiles à interpréter en raison d'un choix conceptuel arrêté par CCP en concertation avec MMOS et les scientifiques, à savoir supprimer les intervalles de chaque côté du graphique. Nous avons fait abstraction de la nomenclature de ces graphiques, d'une part pour lutter contre le botting et d'autre part pour simplifier l'expérience utilisateur. Ce choix a entraîné la suppression des repères intervallaires malgré leur importance, dans la mesure où tous les graphiques que nous avons reçus s'inscrivent dans une échelle logarithmique. Cela signifie que plus le chiffre correspondant à l'intervalle augmente, plus le nombre de protéines présentes dans cette partie de la cellule croît exponentiellement. Un point équivaut à 1 000 protéines sur le repère intervallaire étiqueté « 3 » sur l'échantillon de Type A. Fondamentalement, un point situé dans l'aire en haut à droite du graphique correspond en quantité de protéines à des milliers de points dans l'aire en bas à gauche du graphique.
Comment cela impacte-t-il la délimitation de clusters par les capsuliers ? Simplement dit, vous devrez désormais vous assurer de séparer les traces des clusters de haute densité. Au lieu de tracer des graphiques qui ressemblent à ça…
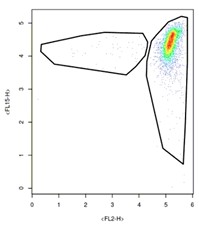
…nous devons créer des graphiques qui s'apparentent à ceci :
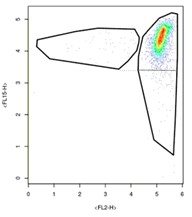
Le procédé est relativement simple pour les graphiques où la trace latérale est alignée sur le cluster de haute densité comme ci-dessus. Dans ce cas, délimitez juste la trace inférieure en alignant votre polygone sur la partie inférieure de la trace opposée. Essayez de délimiter symétriquement vos clusters. La tâche se complique pour les échantillons comme celui-ci :
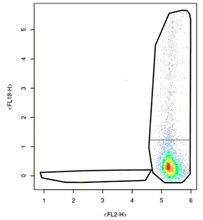
Dans cet échantillon, la trace latérale dans l'aire gauche est bien plus petite que l'autre et entaillerait le cluster de haute densité si elle était délimitée symétriquement. Vous devrez procéder autrement. Traitez plutôt ces échantillons comme si vous aviez affaire à des comètes et que vous essayiez de séparer la queue du corps. Cette délimitation supplémentaire permettra de recueillir des données encore plus précieuses au sujet de ces populations cellulaires uniques.
Outre les graphiques en comète, il est également important de détailler le procédé de délimitation des graphiques de Type B. Comme nous l'avons vu plus haut, ces graphiques cytométriques ressemblent à un amalgame de populations cellulaires. Jusqu'à présent, les capsuliers ont admirablement réussi à délimiter les aires gauche, droite et centrale des graphiques de Type B. Cependant, vous remarquerez la présence d'un cluster de points de données qui semble se diffuser hors de la zone de haute densité. Pensez à ces points comme des cellules renégates, luttant pour se libérer du cluster principal. Ces points doivent être catégorisés comme un groupe à part entière.
Pour ce faire, segmentez le polygone au niveau du point de séparation le plus mince entre ces deux clusters. Lorsqu'aucune délimitation n'apparaît clairement, essayez de faire concorder symétriquement chaque cluster, puis de tracer une ligne de séparation à l'intersection des deux clusters.
Quoi qu'il en soit, la quantité inouïe d'échantillons fiables fournis jusqu'à aujourd'hui par les joueurs d'EVE Online est prodigieuse. Il ne fait aucun doute que vous relèverez haut la main le défi de délimiter encore plus précisément les ensembles de données les plus complexes du Project Discovery.
« Ce projet dépasse toutes mes attentes : non seulement les joueurs continuent de s'investir sans relâche et montrent un réel intérêt pour nos travaux, mais ils recueillent également une quantité phénoménale de données d'excellente qualité pour nos recherches. Leurs efforts contribueront certes à une meilleure compréhension de la COVID-19, mais les données qu'ils génèrent seront par ailleurs librement et largement partagées au sein de toute la communauté scientifique. En outre, l'exploitation des résultats de leurs analyses suscite un vif intérêt dans le domaine de la génération d'algorithmes d'apprentissage automatique. Il n'existe tout simplement aucune autre ressource comparable à celle actuellement générée. » - Dr Ryan Brinkman, Professeur en Génétique médicale, Université de la Colombie-Britannique, Scientifique émérite à BC Cancer.
